内转录间隔区系统发育分型
内转录间隔区系统发育分型
内转录间隔区(ITS)是核糖体转录的区域,在成熟过程中会被切除并降解。它们的序列通常比核糖体序列显示出更多的变异,从而使其广泛用于系统发育分析和/或鉴定物种和菌株。尤其对于真菌而言,这是一种广泛使用的鉴定技术,因为基于形态特征进行鉴定很费力,而且通常不会得到正确的结果。所使用的区域通常是ITS和核糖体序列的组合,并且通常由(部分)18S rRNA序列(原核生物为16S rRNA)、内转录间隔区(ITS1)、5.8s rRNA的整个序列、内转录间隔区(ITS2)和28s rRNA的(部分)序列组成。在公共数据库(例如NCBI和EBI)上可以找到许多ITS序列。
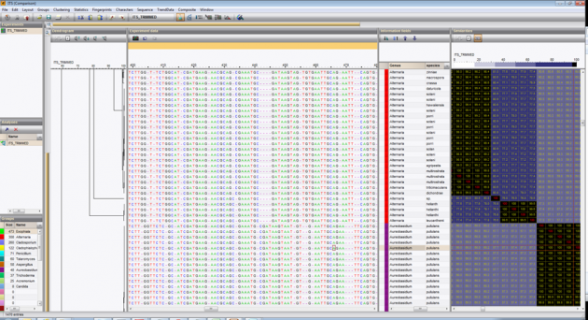
Bionumerics软件中ITS的分析流程
在BioNumerics中,可以使用批处理序列拼接插件从跟踪文件中批量导入ITS序列。可以从公共数据库和/或fasta或GenBank文件中导入参考序列。
Needleman-Wunsch,Wilbur-Lipman和Bionumerics的专有算法可以使用多种算法对序列进行多重序列比对。可以人工调整比对参数设置。基于多重序列比对,可以进行聚类以反映所分析序列之间的系统发育关系。
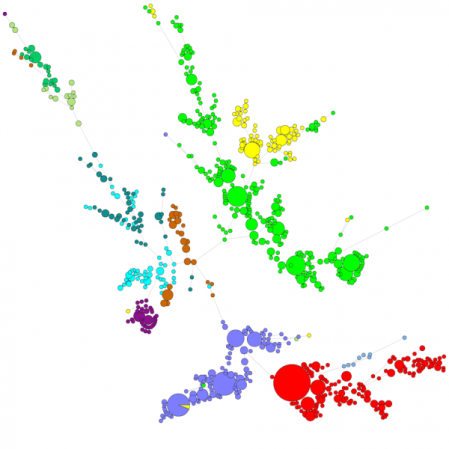
当比较大量序列时,UPGMA或邻位相邻的树状图不能很好地概述数据。在这些情况下,无根树提供了更好的解决方案,在BioNumerics中,可以从序列数据开始计算最小生成树、最大似然树和最大简约树。可以为数据定义不同的组,这些组在树中可视化展示,从而很好地概述了系统发育关系、数据库中存在的聚类以及异常值。下图显示了基于ITS序列的最小生成树,各组代表不同的属。
ITS序列还可用于根据数据库识别未知生物。 使用分类鉴定模块,用户可以创建包含已识别菌株的识别项目,并使用这些项目来识别未知菌株,将基于相似度以及评分的可靠性来分配得分。用户可以完全自定义用于比较的参数和用于鉴定的临界值。
以上资料由BioNumerics中国区官方授权总经销商独家提供,如需转载请主动标明出处。





